In un precedente articolo su questo blog sono stati esposti un metodo geometrico ed un metodo analitico per la costruzione/determinazione di una molecola in tassellazioni "flagstone".
In particolare il metodo geometrico permette agevolmente, attraverso programmi di grafica vettoriale, di costruire una molecola flagstone di forma e dimensioni specifiche.
Tuttavia, se si vuole progettare una intera tassellazione flagstone, formata ad esempio da molecole flagstone differenti, allora l'applicazione del metodo comincia a diventare dispendiosa in termini di tempo,
per via del crescente numero di passaggi ripetitivi.
Da qui è nata l'esigenza di uno strumento che permettesse l'automatizzazione di questi passaggi e fornisse direttamente il crease pattern finale della molecola desiderata;
ossia il software OriFlagTess.
Il software
OriFlagTess è un software libero sviluppato in C# sotto ambiente Microsoft Windows.
Il "core" del programma si basa principalmente sul calcolo analitico mostrato nell'articolo sopra citato;
in aggiunta vengono utilizzate le formule analitiche di traslazione e rotazione per la rappresentazione finale a video della molecola.
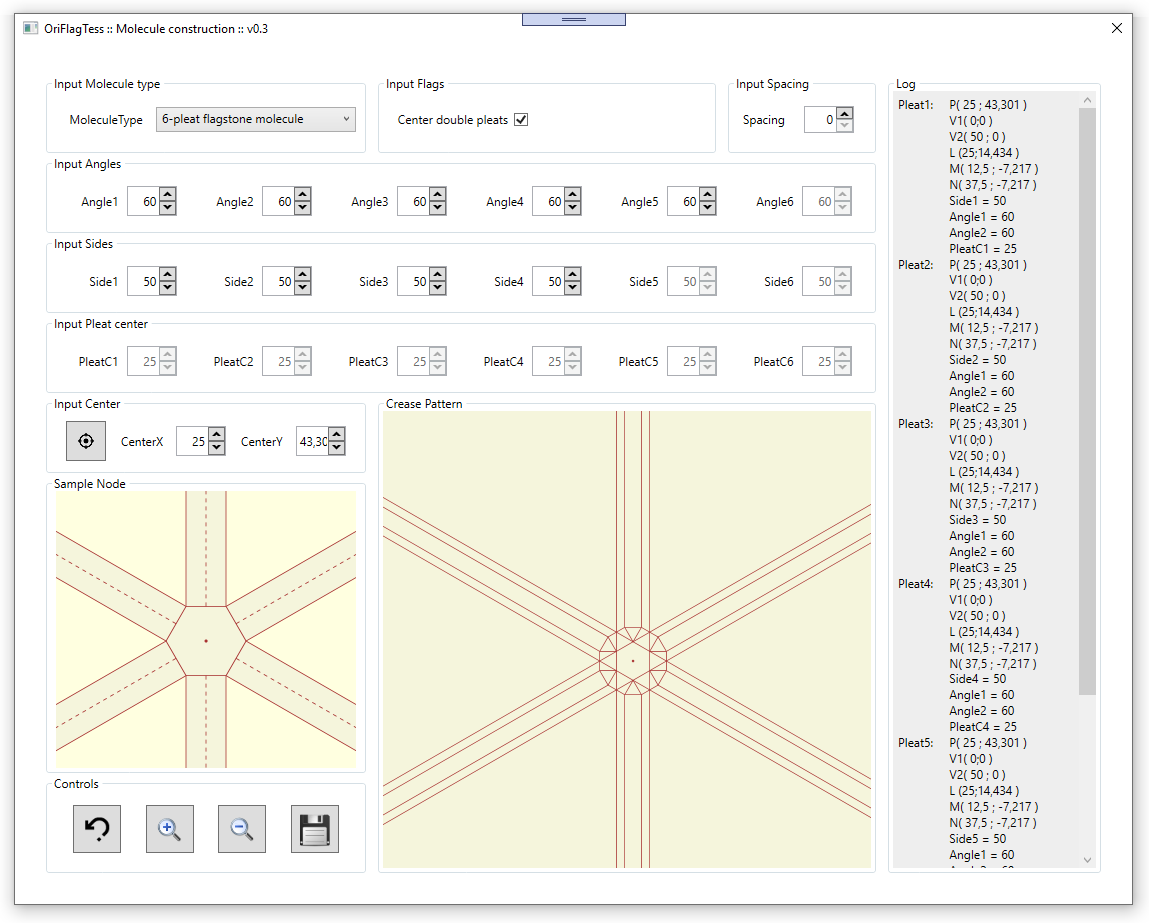
Il software consiste in un'unica finestra principale in cui è possibile inserire tutti i valori di input, e visualizzare in tempo reale il risultato,
ossia il crease pattern della molecola desiderata.
I valori che è possibile specificare in input sono:
- Tipo della molecola
- le coordinate del centro della molecola
- la lunghezza dei lati della molecola (espressa in pixel) 12
- gli angoli della molecola (espressi in gradi) 34
- i punti di ciascun lato in cui si vuole far convergere la relativa doppia piega (espressi come distanza da un estremo del lato) ("pleat center")
- la spaziatura desiderata tra le doppie pieghe nel modello finale (la "fuga" della molecola)
Ad ogni inserimento/modifica di un valore si ha, in tempo reale, il ricalcolo e la visualizzazione a video del nuovo crease pattern della molecola.
Nella finestra troviamo anche un riquadro di aiuto in cui viene evidenziato in tempo reale l'equivalente del valore che stiamo inserendo/modificando.
Infine, una funzionalità fondamentale è ovviamente la possibilità di salvare in formato vettoriale (SVG) il crease pattern ottenuto come risultato.
Esempio di generazione di una molecola a 5 rami
Supponiamo di voler generare il crease pattern di una molecola flagston a 5 rami.
Eseguiamo il software, e nell'ordine:
- selezioniamo "5-pleat" come tipo di molecola
- inseriamo il valore in gradi dei primi $4$ angoli, ossia $80$, $72$, $80$ e $72$
- inseriamo la lunghezza dei primi $3$ lati, ossia $50$, $60$ e $65$
- inseriamo le coordinate del centro della molecola o clicchiamo sul pulsante "auto center"
A questo punto in tempo reale, abbiamo già ottenuto il crease pattern definitivo della molecola.
Non ci resta che salvarlo cliccando sul pulsante apposito.
Il video di seguito mostra questo esempio appena descritto:
Repository e download
Il software è distribuito liberamente con licenza GPL v3.
E' disponibile su github, all'indirizzo https://github.com/UnclePetros/OriFlagTess/.
A tale indirizzo, nella schermata iniziale, trovate delle brevi istruzioni su come scaricare ed installare il software.
Se volete scaricare direttamente il software eseguibile, cliccate QUI.
Enjoy!
P.S.: per qualsiasi feedback sul software e sul suo utilizzo potete utilizzare i commenti qui sotto, oppure l'indirizzo mail  .
.
Riferimenti bibliografici
- Robert J. Lang, Twists, Tilings, And Tessellations, CRC Press, 2017.
- Bencini, Di Rocco, Geronimo, Il pensiero matematico 1, Ferraro, 1996.
-
Da notare che i lati specificabili in input, sono solo i primi $n-2$ partendo dal basso e proseguendo in senso antiorario, dove $n$ è il numero di rami della molecola. I restanti $2$ lati vengono determinati automaticamente per costruzione. ↩
-
i valori inseriti corrisponderanno ad altrettanti pixel nell'immagine svg che si andrà a salvare al termine. ↩
-
si fa riferimento agli angoli "esterni" della molecola, ossia gli angoli che andranno a formare il nodo finale, al termine della piegatura ↩
-
gli angoli specificabili in input sono solo i primi $n-1$ partendo da quello in basso a sinistra e proseguendo in senso antiorario; l'ultimo angolo viene determinato automaticamente per differenza da $360°$ ↩






